
ابزار هوش مصنوعی قوانین مهندسی پروتئین را از نو مینویسد
به گزارش گروه دانشگاه خبرگزاری دانشجو، گروهی از دانشمندان در چین، به رهبری پروفسور کایشیا گائو از موسسه ژنتیک و زیستشناسی تکوینی (IGDB) در آکادمی علوم چین، روشی نوآورانه را معرفی کردهاند که ممکن است مهندسی پروتئین را به طور قابل توجهی پیشرفت دهد. این تکنیک جدید، که به عنوان محدودیتهای آگاهانه هوش مصنوعی برای مهندسی پروتئین (AiCE) شناخته میشود، با ترکیب بینشهای ساختاری و تکاملی در یک مدل استاندارد تاخوردگی معکوس، تکامل پروتئینها را تسریع میکند. نکته قابل توجه این است که این کار بدون نیاز به توسعه یا آموزش سیستمهای هوش مصنوعی اختصاصی (AI) انجام میشود.
این تحقیق که در تاریخ ۷ جولای در مجله Cell منتشر شد، چندین محدودیت دیرینه در روشهای مرسوم مهندسی پروتئین را هدف قرار میدهد.
در حالت ایدهآل، مهندسی پروتئین میتواند با حداقل پیچیدگی به نتایج با کارایی بالا دست یابد. با این حال، اکثر روشهای فعلی با هزینههای بالا، راندمان پایین و مقیاسپذیری محدود دست و پنجه نرم میکنند. اگرچه رویکردهای مبتنی بر هوش مصنوعی پیشرفتهایی را ارائه میدهند، اما معمولاً به منابع محاسباتی قابل توجهی نیاز دارند. این امر نیاز به راهحلهایی را ایجاد کرده است که هم قدرتمند و هم کاربردی باشند و امکان استفاده گستردهتر را بدون کاهش دقت فراهم کنند.
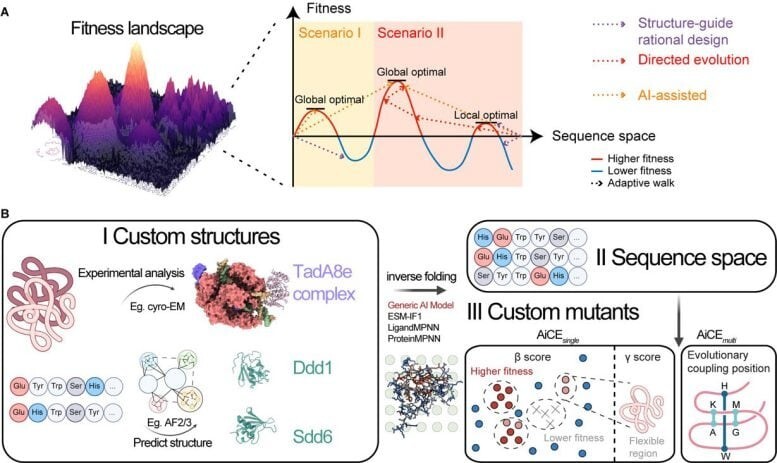
در این مطالعه، محققان ابتدا AiCE single را توسعه دادند، ماژولی که برای پیشبینی جایگزینیهای تک اسید آمینه با تناسب بالا (HF) طراحی شده است. این ماژول با نمونهبرداری گسترده از مدلهای تاخوردگی معکوس - مدلهای هوش مصنوعی که توالیهای اسید آمینه سازگار را بر اساس ساختارهای سهبعدی پروتئین تولید میکنند - ضمن در نظر گرفتن محدودیتهای ساختاری، دقت پیشبینی را افزایش میدهد.
رویکرد آگاهانه هوش مصنوعی AiCE برای مهندسی پروتئین
مقایسه با ۶۰ مجموعه داده اسکن جهش عمیق (DMS) نشان داد که روش AiCE single از سایر روشهای مبتنی بر هوش مصنوعی بین ۳۶ تا ۹۰ درصد بهتر عمل میکند. اثربخشی آن برای پروتئینهای پیچیده و کمپلکسهای پروتئین-اسید نوکلئیک نیز تأیید شد. نکته قابل توجه این است که در نظر گرفتن محدودیتهای ساختاری به تنهایی باعث بهبود ۳۷ درصدی در دقت شد.
برای پرداختن به چالش برهمکنشهای اپیستاتیک منفی در جهشهای ترکیبی، محققان ماژول چندگانه AiCE را توسعه دادند که محدودیتهای جفت شدن تکاملی را ادغام میکند. این امر امکان پیشبینی دقیق جهشهای چندگانه با تناسب بالا را با حداقل هزینه محاسباتی فراهم میکند و تطبیقپذیری و کاربرد عملی ابزار را گسترش میدهد.
مهندسی نسل جدیدی از پروتئینهای عملکردی
محققان با استفاده از چارچوب AiCE، با موفقیت هشت پروتئین با ساختارها و عملکردهای متنوع، از جمله دآمینازها، توالیهای محلیسازی هستهای، نوکلئازها و رونوشتبردارهای معکوس، را تکامل دادند. این پروتئینهای مهندسیشده، امکان ایجاد چندین ویرایشگر باز نسل بعدی را برای کاربردهای پزشکی دقیق و اصلاح مولکولی فراهم کردهاند. این موارد عبارتند از: enABE۸e، یک ویرایشگر باز سیتوزین با پنجره ویرایش تقریباً ۵۰٪ باریکتر؛ enSdd۶-CBE، یک ویرایشگر باز آدنین با ۱.۳ برابر دقت بالاتر؛ و enDdd۱-DdCBE، یک ویرایشگر باز میتوکندری که افزایش ۱۳ برابری در فعالیت را نشان میدهد.
AiCE یک استراتژی ساده، کارآمد و با کاربرد گسترده برای مهندسی پروتئین ارائه میدهد. با آزادسازی پتانسیل مدلهای هوش مصنوعی موجود، مسیر جدید و امیدوارکنندهای را برای این حوزه ارائه میدهد و قابلیت تفسیر طراحی مجدد پروتئین مبتنی بر هوش مصنوعی را افزایش میدهد.
مرجع: «پیشرفت تکامل پروتئین با مدلهای تاشو معکوس با ادغام محدودیتهای ساختاری و تکاملی» توسط Hongyuan Fei، Yunjia Li، Yijing Liu، Jingjing Wei، Aojie Chen و Caixia Gao، ۷ ژوئیه ۲۰۲۵، سلول.
DOI: ۱۰.۱۰۱۶/j.cell.۲۰۲۵.۰۶.۰۱۴

